Weltweit erstes Institut für das Erkennen von Bakterien akkreditiert
Das Institut für Infektionskrankheiten (IFIK) der Universität Bern ist das weltweit erste akkreditierte Labor, welches Bakterien mittels der sogenannten Nanoporen-Sequenzierung identifizieren kann.
Als weltweit erster Anbieter hat das Institut für Infektionskrankheiten (IFIK) der Universität Bern seit 21. Januar 2019 den Akkreditierungsstatus ISO/IEC 17025:2018 erhalten, um spezifische DNA-Fragmente aus Bakterienkulturen mit Hilfe der Oxford Nanopore Technologies in der medizinischen Diagnostik zu sequenzieren.
Diese neue Sequenziertechnologie ermöglicht es, Krankheitserreger schneller und kostengünstig zu identifizieren. Das IFIK-Sequenzierlabor hat Mitte Oktober 2018 auch die Qualifizierung zum zertifizierten Nanopore Service Provider erhalten, die erste in der Schweiz. Dr. Alban Ramette, Gruppenleiter Bioinformatics/Biostatistics, erklärt «uniaktuell», wie die Technologie funktioniert und wofür er sie nutzt.

Was bedeutet die Akkreditierung für Ihr Institut für Infektionskrankheiten (IFIK) der Universität Bern?
Alban Ramette: Wir erachten es als einen grossen Erfolg, das weltweit erste Labor zu sein, welches die Sequenzierung spezifischer DNA-Fragmente mit Geräten unseres Partners, Oxford Nanopore Technologies, akkreditieren konnte. Diese Anerkennung bestätigt die Universität Bern als Kompetenzzentrum für Gesundheitswissenschaften und das IFIK als eine Institution, die wegweisende technologische Innovation effizient in medizinische Diagnostikanwendungen umsetzt.
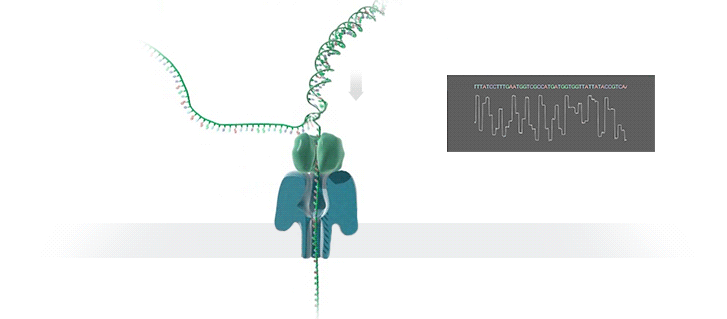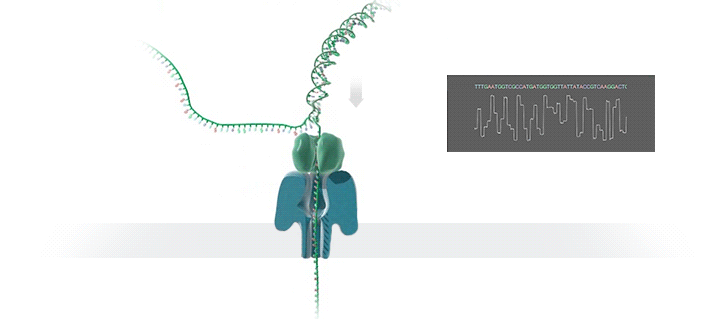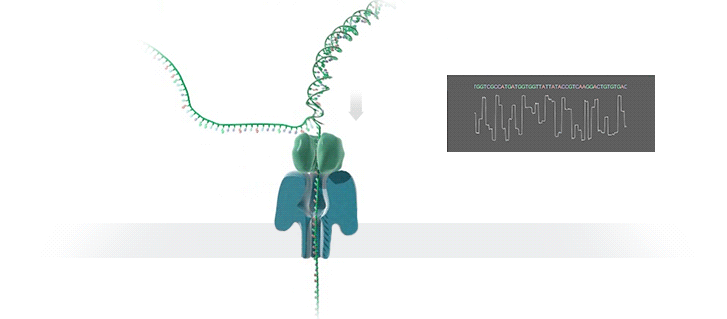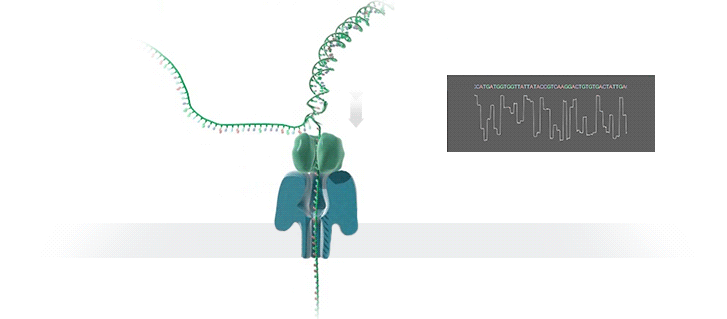
Was ist der Vorteil der neuen Technologie, und wie funktioniert sie genau?
Die neue Technologie ermöglicht eine schnelle Sequenzierung von DNA-Fragmenten und ist ideal für Analysen, bei denen eine hohe Flexibilität bezüglich Probenanzahl und rascher Resultatverfügbarkeit erforderlich ist. Das Prinzip des GridION-Geräts basiert auf der Messung eines Stromflusses beim Durchtritt von biologischen Molekülen durch Nanoporen – das sind Poren im Nanometerbereich in einer Membran. Diese Stromänderungen werden aufgezeichnet und zur Identifizierung der Zusammensetzung der Moleküle verwendet. Mit dieser Technologie können Moleküle unterschiedlicher Herkunft – zum Beispiel aus einer Mischung von pathogenen und unschädlichen Bakterien – gleichzeitig analysiert und durch den Abgleich mit biologischen Referenzdatenbanken identifiziert werden.
Wo kommt die Technologie zum Einsatz?
Wenn ein Erreger aus einer Patientenprobe isoliert wird, sequenzieren wir DNA-Abschnitte – sogenannte genetische Marker – welche taxonomische, also Klassifikations-Informationen über den Krankheitserreger liefern. Der Vergleich dieser Signatursequenzen mit bekannten Referenzsequenzen ermöglicht eine genaue Identifizierung der gefundenen Mikroorganismen. Diese Informationen werden mit den Ergebnissen anderer diagnostischer Tests kombiniert, um eine genaue Identifizierung der Krankheitserreger zu erhalten. Diese Informationen sind für eine gezielte Therapie unerlässlich.
Wer profitiert davon?
Eine schnelle und zuverlässige Analyse von Patienten-Proben, die zur klinischen Diagnostik an das IFIK geschickt werden, ist von entscheidender Bedeutung, um den behandelnden Ärztinnen und Ärzten die bestmöglichen Informationen zur Verfügung zu stellen. In erster Linie kommt diese technologische Innovation also den Patientinnen und Patienten zugute.
Zur Person

Dr. Alban Ramette ist Leiter der Bioinformatics/Biostatistics group am Institut für Infektionskrankheiten (IFIK) der Universität Bern.
Kontakt:
Dr. Alban Ramette, PhD
Leiter Bioinformatics/Biostatistics group
Institut für Infektionskrankheiten, Universität Bern
Tel.: +41 31 632 95 40 / alban.ramette@ifik.unibe.ch
Das Institut für Infektionskrankheiten (IFIK)
Das Institut für Infektionskrankheiten kombiniert Dienstleistungen, Lehre und Forschung unter einem Dach und deckt die gesamte Breite der Mikrobiologischen Diagnostik ab. Als einziges universitäres Institut in der Schweiz vereint es in der Lehre, der Forschung und der Diagnostik alle mikrobiologischen Spezialdisziplinen (wie die Parasitologie, Bakteriologie oder Virologie) unter einem Dach. Zu den Forschungsschwerpunkten des IFIK gehört die Frage, wie sich Antibiotikaresistenzen und Infektionen im Zentralen Nervensystem entwickeln sowie die Gesundheit und Infektionen von Darmbakterien.
Zur Autorin
Nathalie Matter arbeitet als Redaktorin bei Media Relations und ist Themenverantwortliche «Gesundheit und Medizin» in der Abteilung Kommunikation & Marketing an der Universität Bern.